Revista Alergia México Volumen 62, Núm. 2, abril-junio, 2014
Recibido: 18 de noviembre 2014
Aceptado: 27 de enero 2015
Este artículo debe citarse como: Murata C, Ramírez AB, Ramírez G, Cruz A y col. Análisis discriminante para predecir el diagnóstico clínico de inmunodeficiencias primarias: reporte preliminar. Revista Alergia Méx 2015;62:125-133.
Discriminant analysis to predict the clinical diagnosis of primary immunodeficiencies: a preliminary report
Chiharu Murata,1 Ana Belén Ramírez,2 Guadalupe Ramírez,3 Alonso Cruz,3 José Luis Morales,4 Saul Oswaldo Lugo-Reyes,2
1Departamento de Metodología de la Investigación.
2Unidad de Investigación en Inmunodeficiencias.
3Servicio de Inmunología Clínica.
Instituto Nacional de Pediatría, Secretaría de Salud, México, DF.
4Departamento de Matemáticas, Instituto Tecnológico Autónomo de México, Distrito Federal.
Correspondencia: Dr. Saúl O Lugo Reyes. Unidad de Investigación en Inmunodeficiencias , Instituto Nacional de Pediatría, Av. del Imán 1, Torre de Investigación, piso 9, 04530 México, DF. dr.lugo.reyes@gmail.com
Antecedentes: las características clínicas de un paciente con sospecha de inmunodeficiencia primaria orientan el diagnóstico diferencial por medio del reconocimiento de patrones. Las inmunodeficiencias primarias son un grupo heterogéneo de más de 250 enfermedades congénitas con mayor susceptibilidad a padecer infecciones, autoinflamación, autoinmunidad, alergia y cáncer. El análisis discriminante lineal es un método multivariante de clasificación supervisada para agrupar a los sujetos a partir de encontrar combinaciones lineales de un número de variables.
Objetivo: identificar las características que mejor explican la pertenencia de pacientes pediátricos con inmunodeficiencias primarias a un grupo de defectos o a una enfermedad.
Material y método: estudio analítico transversal en el que a partir de una base de datos preexistente, con registros clínicos y de laboratorio de 168 pacientes con inmunodeficiencia primaria, seguidos en el Instituto Nacional de Pediatría de 1991 a 2012, construimos modelos discriminantes lineales para explicar la pertenencia de cada paciente a los diferentes grupos de defectos y a las inmunodeficiencias primarias más prevalentes en nuestro registro. Luego de una corrida preliminar se incluyeron únicamente las 30 variables (4 demográficas, 10 clínicas, 10 de laboratorio y 6 gérmenes) de mayor peso, a partir de las que se construyeron los modelos de entrenamiento con el algoritmo paso-a-paso (stepwise) hacia atrás, utilizando selección automatizada de variables e incorporación manual “teórica” por un experto humano. Se evaluó la utilidad clínica de los modelos resultantes (sensibilidad, especificidad, exactitud y coeficiente kappa), con intervalos de confianza de 95%.
Resultados: los modelos incluyeron 6 a 14 variables para explicar la pertenencia de 168 pacientes con inmunodeficiencias primarias a los cinco grupos más numerosos (combinados, anticuerpos, bien definidos, desregulación y fagocitosis) y las cuatro enfermedades más prevalentes (agammaglobulinemia ligada al cromosoma X, enfermedad granulomatosa crónica, inmunodeficiencia común variable y ataxia-telangiectasia). Prácticamente en todos los casos el desempeño de la máquina fue superior al del experto humano en lo que respecta a la selección de los atributos más pertinentes para incorporar en los modelos. La predicción del diagnóstico con base en las ecuaciones construidas tuvo exactitud global de 83 a 94%, con sensibilidad de 60 a 100%, especificidad de 83 a 95% y coeficiente kappa de 0.37 a 0.76.
Conclusiones: la selección de variables, en general, tiene plausibilidad clínica y tiene la ventaja práctica de utilizar solamente atributos clínicos, gérmenes encontrados y estudios de laboratorio de rutina (biometría hemática e inmunoglobulinas séricas). El desempeño del modelo como herramienta de predicción fue aceptable. Las principales limitaciones del estudio incluyen un tamaño de muestra limitado, lo que no permitió que realizáramos validación cruzada en la evaluación. Éste es solamente un primer paso en la construcción de un sistema de aprendizaje automático, con un abordaje más amplio que incluya una base de datos más grande y diferentes metodologías, para asistir el diagnóstico clínico de las inmunodeficiencias primarias.
Palabras clave: análisis discriminante, diagnóstico clínico, inmunodeficiencias primarias, aprendizaje automático, asistido por computadora, experto vs máquina.
Background: The features in a clinical history from a patient with suspected primary immunodeficiency (PID) direct the differential diagnosis through pattern recognition. PIDs are a heterogeneous group of more than 250 congenital diseases with increased susceptibility to infection, inflammation, autoimmunity, allergy and malignancy. Linear discriminant analysis (LDA) is a multivariate supervised classification method to sort objects of study into groups by finding linear combinations of a number of variables.
Objective: To identify the features that best explain membership of pediatric PID patients to a group of defect or disease.
Material and method: An analytic cross-sectional study was done with a pre-existing database with clinical and laboratory records from 168 patients with PID, followed at the National Institute of Pediatrics during 1991-2012, it was used to build linear discriminant models that would explain membership of each patient to the different group defects and to the most prevalent PIDs in our registry. After a preliminary run only 30 features were included (4 demographic, 10 clinical, 10 laboratory, 6 germs), with which the training models were developed through a stepwise regression algorithm. We compared the automatic feature selection with a selection made by a human expert, and then assessed the diagnostic usefulness of the resulting models (sensitivity, specificity, prediction accuracy and kappa coefficient), with 95% confidence intervals.
Results: The models incorporated 6 to 14 features to explain membership of PID patients to the five most abundant defect groups (combined, antibody, well-defined, dysregulation and phagocytosis), and to the four most prevalent PID diseases (X-linked agammaglobulinemia, chronic granulomatous disease, common variable immunodeficiency and ataxia-telangiectasia). In practically all cases of feature selection the machine outperformed the human expert. Diagnosis prediction using the equations created had a global accuracy of 83 to 94%, with sensitivity of 60 to 100%, specificity of 83 to 95% and kappa coefficient of 0.37 to 0.76.
Conclusions: In general, the selection of features has clinical plausibility, and the practical advantage of utilizing only clinical attributes, infecting germs and routine lab results (blood cell counts and serum immunoglobulins). The performance of the model as a diagnostic tool was acceptable. The study’s main limitations are a limited sample size and a lack of cross validation. This is only the first step in the construction of a machine learning system, with a wider approach that includes a larger database and different methodologies, to assist the clinical diagnosis of primary immunodeficiencies.
Key words: discriminant analysis, clinical diagnosis, primary immunodeficiencies, automatic learning, computed-assised, expert vs machine.
Las inmunodeficiencias primarias son un grupo heterogéneo de más de 200 enfermedades genéticas con incidencia global aproximada de 1 en 10,000 recién nacidos vivos.1 Su manifestación clínica es variable, la mayor parte de ellas confiere más susceptibilidad a gérmenes y se manifiestan principalmente como infecciones inusuales, graves, persistentes o de repetición. Los antecedentes familiares, la consanguinidad, la edad de inicio, el sexo, los sitios de infección, las manifestaciones asociadas, las complicaciones, los hallazgos de laboratorio y los gérmenes aislados son características a tomar en cuenta para orientar el diagnóstico clínico de las inmunodeficiencias primarias.
El diagnóstico clínico incluye la selección de atributos relevantes y el reconocimiento de patrones,2,3 un proceso complejo que requiere contemplar varias combinaciones de variables y para la mente humana puede ser difícil cubrir todas las posibilidades;4 una herramienta computacional que genere el diagnóstico diferencial automatizado basado en modelos estadísticos puede complementar y facilitar esta tarea.
Uno de los modelos estadísticos que se utilizan para ayudar a establecer diagnósticos de enfermedades es el discriminante lineal. La meta de este método es clasificar correctamente la pertenencia de un elemento a un grupo con base en la función discriminante. Se entrena el modelo con casos disponibles, de los que se conoce la pertenencia a categorías de interés, junto con los atributos que serán elegidos para la construcción del modelo. Con la función discriminante se calcula la probabilidad de un nuevo caso problema de pertenecer a una categoría específica.5
En Medicina, el modelo discriminante lineal se ha utilizado principalmente en la evaluación de ensayos clínicos con múltiples variables de desenlace y en la selección de atributos para la reducción de dimensionalidad. En el campo de diagnóstico médico se conocen como antecedentes exitosos: la distinción temprana entre trastorno bipolar y esquizofrenia en adolescentes,6 procesamiento de electroencefalogramas para diagnosticar autismo7 y la clasificación de tejidos con base en expresión génica,8 entre otros.5
En la actualidad existen más de 250 inmunodeficiencias primarias identificadas y se estima que para el año 2020 alcancen más de 2,000. La cantidad de expertos en inmunodeficiencias primarias es escasa en México y el mundo, por lo que consideramos útil el desarrollo de un sistema de aprendizaje automático que pueda optimizar y facilitar el proceso diagnóstico y que sea accesible para médicos de otras especialidades y en hospitales apartados geográficamente.
En este estudio reportamos los resultados preliminares de un proyecto en proceso de realización, cuyo objetivo es obtener modelos discriminantes lineales para establecer el diagnóstico de cinco tipos de inmunodeficiencias primarias y cuatro enfermedades de inmunodeficiencias primarias de frecuencia relativamente alta, evaluar la utilidad de la prueba diagnóstica de estos modelos, así como compararlos con los modelos constituidos mediante las variables seleccionadas por un médico inmunólogo pediatra experto en inmunodeficiencias primarias.
Estudio analítico transversal en el que usamos una base de datos con registros clínicos y de laboratorio de 168 pacientes pediátricos de casos atendidos y en seguimiento en el Instituto Nacional de Pediatría entre 1991 y marzo de 2012, para construir los modelos discriminantes, con el fin de predecir la pertenencia de un paciente a uno de cinco grupos de defectos (celulares o combinados [Grupo I], predominantemente de anticuerpos [Grupo II], síndromes bien definidos [Grupo III], desregulación inmunitaria [Grupo IV] y defectos de la fagocitosis [Grupo V]), y a una de cuatro enfermedades (agammaglobulinemia ligada al cromosoma X, inmunodeficiencia común variable, ataxia-telangiectasia y enfermedad granulomatosa crónica). Los diagnósticos los establecieron médicos especialistas del Servicio de Inmunología clínica del Instituto Nacional de Pediatría, con base en una historia clínica compatible y estudios de laboratorio y moleculares, de acuerdo con criterios internacionales.
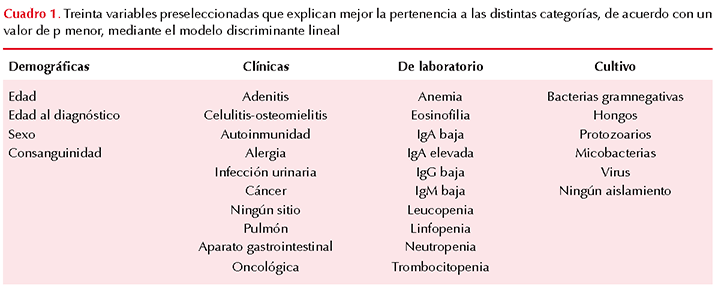
Se incluyeron variables demográficas, antecedentes familiares, de infecciones, manifestaciones asociadas y complicaciones, aislamiento de microorganismos infectantes y hallazgos de laboratorios, incluidos éstos solamente en la citometría hemática y las concentraciones séricas de inmunoglobulinas (IgG, IgA, IgM). Por medio de una corrida de filtración de atributos, se seleccionaron 30 variables (4 demográficas, 10 clínicas, 10 de laboratorio y 6 de cultivo) con base en un valor menor de p, para incluir en modelos de entrenamiento del modelo discriminante lineal (Cuadro 1). La selección de variables se realizó con el algoritmo de stepwise hacia atrás, instalado en el paquete estadístico JMP11 de SAS Institute, Inc.; las categorías de referencia fueron el diagnóstico establecido por los médicos mencionados.

De manera independiente, un médico inmunólogo, experto en inmunodeficiencias primarias en población pediátrica, seleccionó variables de importancia teórica entre las mismas 30 variables que se utilizaron para obtener modelos discriminantes, para cada uno de los cinco grupos y cuatro enfermedades de inmunodeficiencias primarias. Los modelos construidos por medio de la selección de variables automatizada y la selección teórica de variables por el médico experto se sometieron a la evaluación de utilidad de prueba diagnóstica, que incluyó: sensibilidad, especificidad, exactitud global y el coeficiente kappa de Cohen.
Se compararon también las variables seleccionadas por diferentes modalidades de la construcción: algoritmo automatizado basado en la función discriminante versus el juicio del experto, así como su número de variables incluidas en el modelo. Los estimadores de parámetros se reportaron con su intervalo de confianza de 95%. Todos los análisis estadísticos se realizaron con el paquete estadístico JMP11 del SAS Institute, Inc.
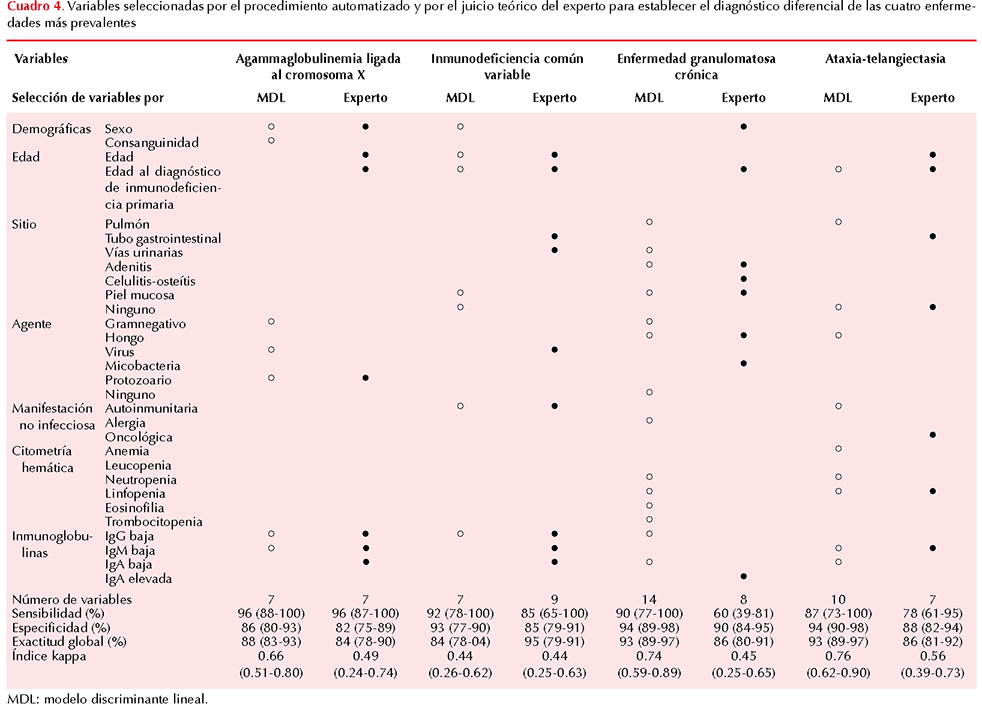
Las características de los pacientes se muestran en el Cuadro 2. Los resultados de la evaluación de la utilidad de la prueba diagnóstica para los cinco grupos y cuatro enfermedades de inmunodeficiencias primarias se muestran en los Cuadros 3 y 4. En la mayor parte de los modelos el procedimiento automatizado tuvo igual o mejor rendimiento en los parámetros de utilidad diagnóstica, comparado con el modelo basado en la selección “teórica” de variables por el experto humano.


El análisis discriminante para el grupo I, Defectos celulares o combinados, identificó como variables pertinentes para el modelo: edad al diagnóstico, infecciones del tubo gastrointestinal, adenitis, manifestaciones alérgicas, leucopenia e IgM baja. También, curiosamente, “ningún sitio de infección” se incluyó en el modelo. Estas siete variables predicen la pertenencia al grupo I en nuestra base de datos con 100% de sensibilidad, 91% de especificidad y 91% de exactitud. La pertenencia al grupo II, Defectos predominantemente de anticuerpos, se explica mediante el modelo discriminante lineal por siete variables: edad al diagnóstico, adenitis, ningún sitio, aislamiento de bacterias gramnegativas, leucopenia, IgG e IgA bajas; con sensibilidad de 78%, especificidad de 95%, exactitud de 89% e índice de concordancia kappa de 0.75. El modelo para explicar la pertenencia al grupo diagnóstico III, Síndromes bien definidos, incorporó ocho variables: edad, edad al diagnóstico, adenitis, hongos, linfopenia, eosinofilia, trombocitopenia e IgG baja. Alcanzó sensibilidad de 83%, especificidad de 78%, exactitud de 80% y coeficiente kappa de 0.55. El análisis discriminante del grupo IV, Desregulación inmunitaria, arrojó como variables relevantes “ningún sitio de infección”, virus y linfopenia; con sensibilidad de 50%, especificidad de 97% y exactitud de 92%, con coeficiente kappa de 0.28. El grupo V, Defectos de la fagocitosis, se explicaría principalmente por nueve variables: infección pulmonar, infección gastrointestinal, celulitis-osteítis, alergia, micobacterias, neutropenia, eosinofilia, trombocitopenia e IgA baja. Los grupos VI, VII y VIII no tuvieron suficientes pacientes para efectuar un análisis de clasificación.
Los diagnósticos más prevalentes en la base de datos incluyeron: agammaglobulinemia ligada al cromosoma X, inmunodeficiencia común variable, ataxia-telangiectasia y enfermedad granulomatosa crónica. Aplicamos el modelo discriminante lineal para explicar y predecir el diagnóstico de pacientes con estas cuatro enfermedades y encontramos: para agammaglobulinemia ligada al cromosoma X, cinco variables: sexo, IgG e IgM bajas, protozoarios y bacterias gramnegativas; con sensibilidad de 92%, especificidad de 86%, exactitud de 87% y coeficiente kappa de 0.62. Para la inmunodeficiencia común variable, dos variables: edad al diagnóstico e IgG baja, con sensibilidad de 85%, especificidad de 82%, exactitud de 82% y coeficiente kappa de 0.37. Para ataxia-telangiectasia, nueve variables: infección en “ningún sitio”, autoinmunidad, cáncer, anemia, neutropenia, linfopenia, IgG, IgM e IgA bajas; la predicción con este modelo alcanzó 87% de sensibilidad, 95% de especificidad, 93% de exactitud y coeficiente kappa de 0.75. Para la enfermedad granulomatosa crónica se incorporaron en el modelo 10 variables: infección pulmonar, infección urinaria, adenitis, celulitis-osteítis, alergia, neutropenia, eosinofilia, trombocitopenia, IgG e IgA bajas; con lo que se alcanzó 90% de sensibilidad, 90% de especificidad, exactitud de 90% y coeficiente kappa de 0.66.
Exploramos una base de datos con registros reales de 168 pacientes pediátricos con diagnóstico conocido de inmunodeficiencias primarias. El análisis automatizado mediante el modelo discriminante lineal permitió identificar varios atributos demográficos, clínicos y de laboratorio que explican la pertenencia de los sujetos al grupo o la enfermedad en la que están clasificados, con desempeño del modelo predictivo muy aceptable, a pesar de que sólo se incorporaron variables clínicas y de laboratorio de rutina (biometría hemática, cultivo e inmunoglobulinas séricas). La sensibilidad, especificidad y exactitud alcanzadas se mantuvieron en los límites de 80 y 98% en la mayoría de los casos. Desde el punto de vista clínico y didáctico, las variables incluidas en los modelos construidos por el procedimiento automatizado mostraron interpretabilidad plausible desde el punto de vista teórico, aunque la discrepancia entre la selección de variables por estos modelos y por el juicio del experto fue importante.
Nuestro estudio tiene varias limitaciones. Primero, debido al tamaño de muestra reducido, sobre todo de algunos grupos y de enfermedad de inmunodeficiencias primarias, en este reporte preliminar no se pudo realizar validación cruzada, que es indispensable para evitar el sobreajuste del modelo para los datos de los que se crearon los mismos modelos. De hecho, se esperaba que entre el diagnóstico de referencia y el diagnóstico generado por el modelo se observaran buenas concordancias. En este punto lo que parece más importante es que, primero, entre los modelos automáticos y modelos teóricos elaborados por el médico experto en inmunodeficiencias primarias se observaron niveles muy similares de los valores de utilidad diagnóstica, y segundo, a pesar de la discrepancia en la selección de variables entre los modelos automático y teórico, en la primera se reconoce una coherencia con la lógica biológica. Asimismo, los niveles de concordancia determinados por medio del coeficiente kappa fueron similares en ambos procedimientos.
Realizar el estudio con un mayor número de casos incorporados en la base de datos permitirá incluir la validación cruzada, con la que esperamos obtener conclusiones más generalizables. En la actualidad estamos en proceso de realizar la construcción de la base de datos que incluirá alrededor de 500 casos y es muy probable que esta nueva base de datos permita generar los resultados con mayor estabilidad y más generalizables.
Hasta donde sabemos, ésta es la primera vez que se aplica el modelo discriminante lineal para predecir el diagnóstico clínico de inmunodeficiencias primarias. Los resultados alentadores de este estudio explorador sugieren la factibilidad de un abordaje más completo y extenso de predicción y asistencia al diagnóstico clínico por medio de métodos de aprendizaje automático. El siguiente paso es, además de incluir la validación cruzada que faltó en este reporte preliminar, probar con otros modelos de aprendizaje automático que muestran buenos rendimientos para la predicción del diagnóstico,9-11 como el modelo de regresión logística, máquinas de soporte vectorial, redes neuronales artificiales, entre otros.
Estos resultados se presentaron durante la reunión semestral de la Asociación de Investigación Pediátrica en Mazatepec, Morelos, 2014, y las observaciones y comentarios ahí vertidos nos permitieron enriquecer este manuscrito.